生物信息软件综合实践实验报告
实验题目 :基因预测和基因结构分析
实验日期:2023年12月5日 星期二
实验者:生物信息2102代子希
实验目的:
1.掌握常用基因从头预测软件的使用和结果解读
2.熟悉文件格式GFF3的基本信息
3.熟悉至少一种基因组浏览器的使用
4.了解基因结构和非编码基因预测等分析
实验内容:
1.使用软件Prodigal预测大肠杆菌Escherichia coli K12基因组的基因,并对预测结果进行总结
2.至少使用两种基因从头预测软件预测拟南芥基因组(部分序列)的基因,并对预测结果进行总结和差异比较。
3.使用IGV查看拟南芥重要基因的注释结果
实验流程和结果
问题1
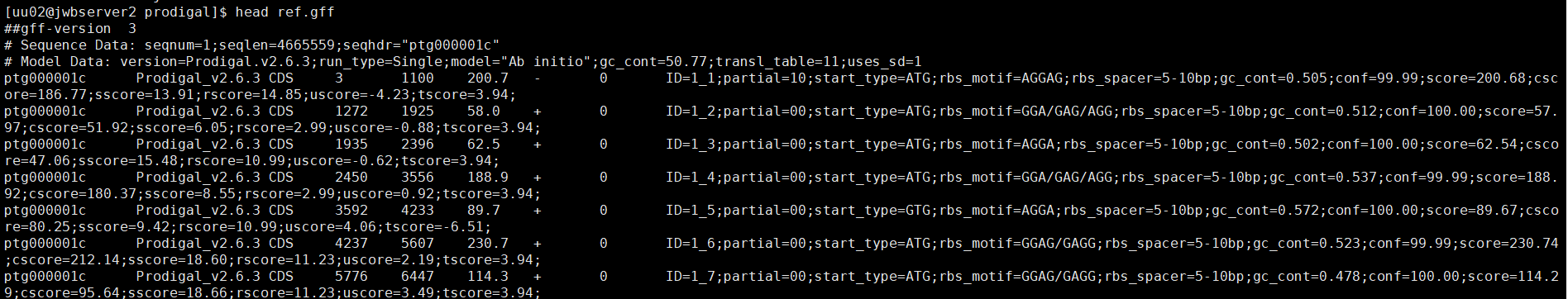
1.使用Prodigal对组装的大肠杆菌的序列(其中1Mb序列,已提供,见文件ecoli.hifi.fa)进行基因预测,统计预测得到的基因的数量
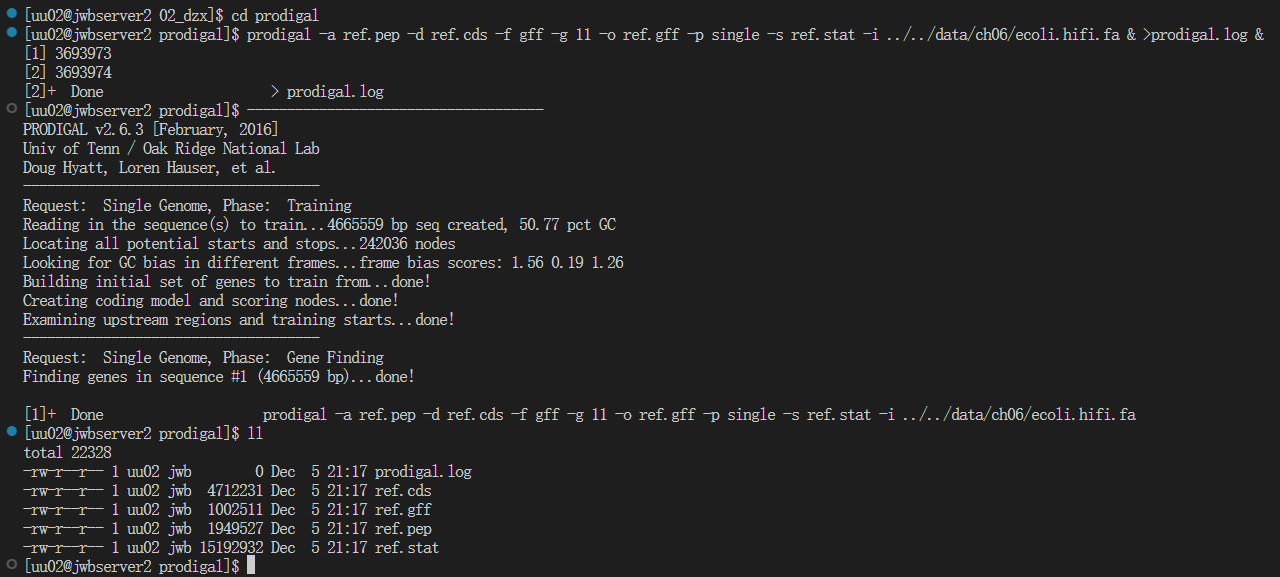
步骤1:使用prodigal命令
1
2
3
|
mkdir prodigal
cd prodigal
prodigal -a ref.pep -d ref.cds -f gff -g 11 -o ref.gff -p single -s ref.stat -i ../../data/ch06/ecoli.hifi.fa & >prodigal.log &
|

ref.cds 输出的预测基因的序列文件
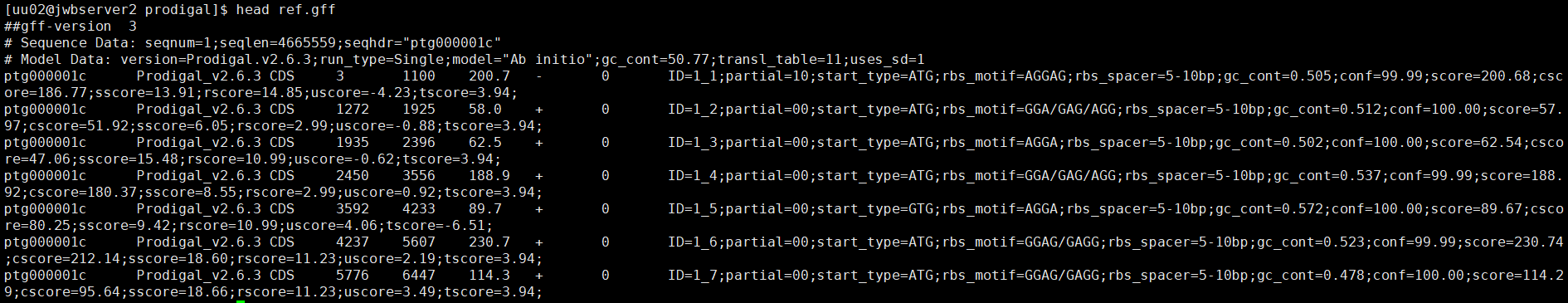
ref.gff 输出的基因的位置信息gff文件
ref.pep 输出的预测序列的氨基酸文件
prodigal文件夹下结果


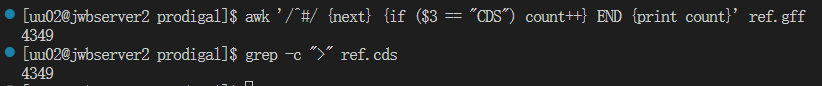
步骤2:统计预测得到的基因的数量
①使用awk命令对gff文件进行统计
1
|
awk '/^#/ {next} {if ($3 == "CDS") count++} END {print count}' ref.gff
|
②使用grep命令对ref.cds文件进行统计

问题2
2.从给定的拟南芥基因组(TAIR10版本)的某段序列(二号染色体7.5-8.5Mb,文件名:Ath.1mb.fa),完成以下任务:
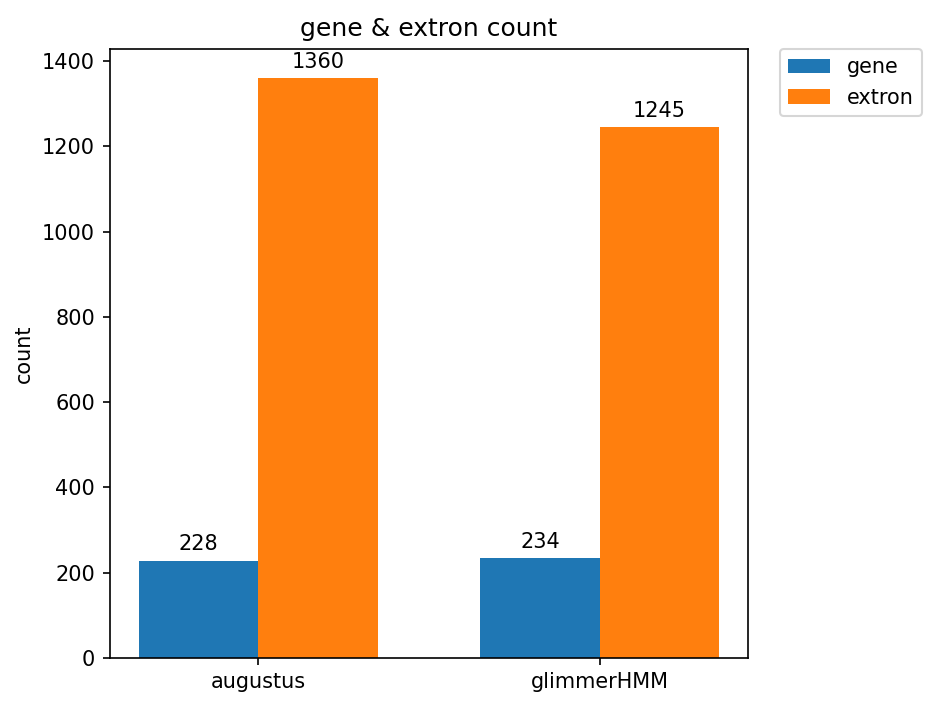
- 使用GenScan、Augustus、GlimmerHMM等软件(至少两种软件)预测该序列所包含的蛋白质编码基因。统计不同软件组装得到的基因和外显子(exon)的数量,并选用适当的图表(如直方图)将统计结果进行展示。
- 从TAIR10网站下载该区间的基因注释信息(已下载到服务器,见文件tair10.ch2_7.5-8.5Mb.genes.gff3),作为标准参考集,试评估第1)题中使用的不同软件预测结果的准确率和召回率等。(可以考虑从基因、exon、CDS等水平上进行比较,比如:对于某个基因,augustus预测的跟tair10的基因重叠区域超过各自注释区间的90%,则认为两者一致,其他exon、CDS的比较,可采用相同标准)
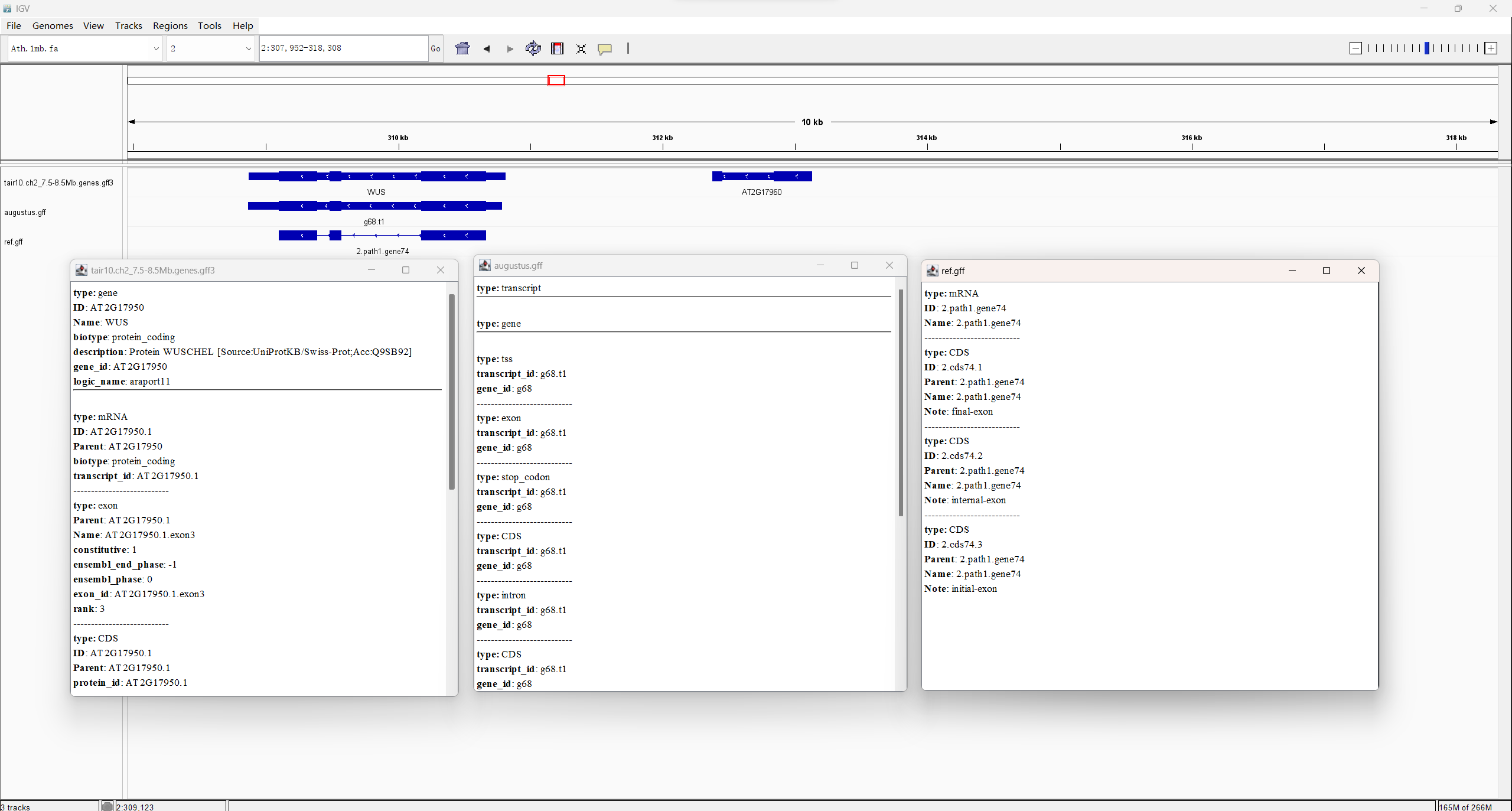
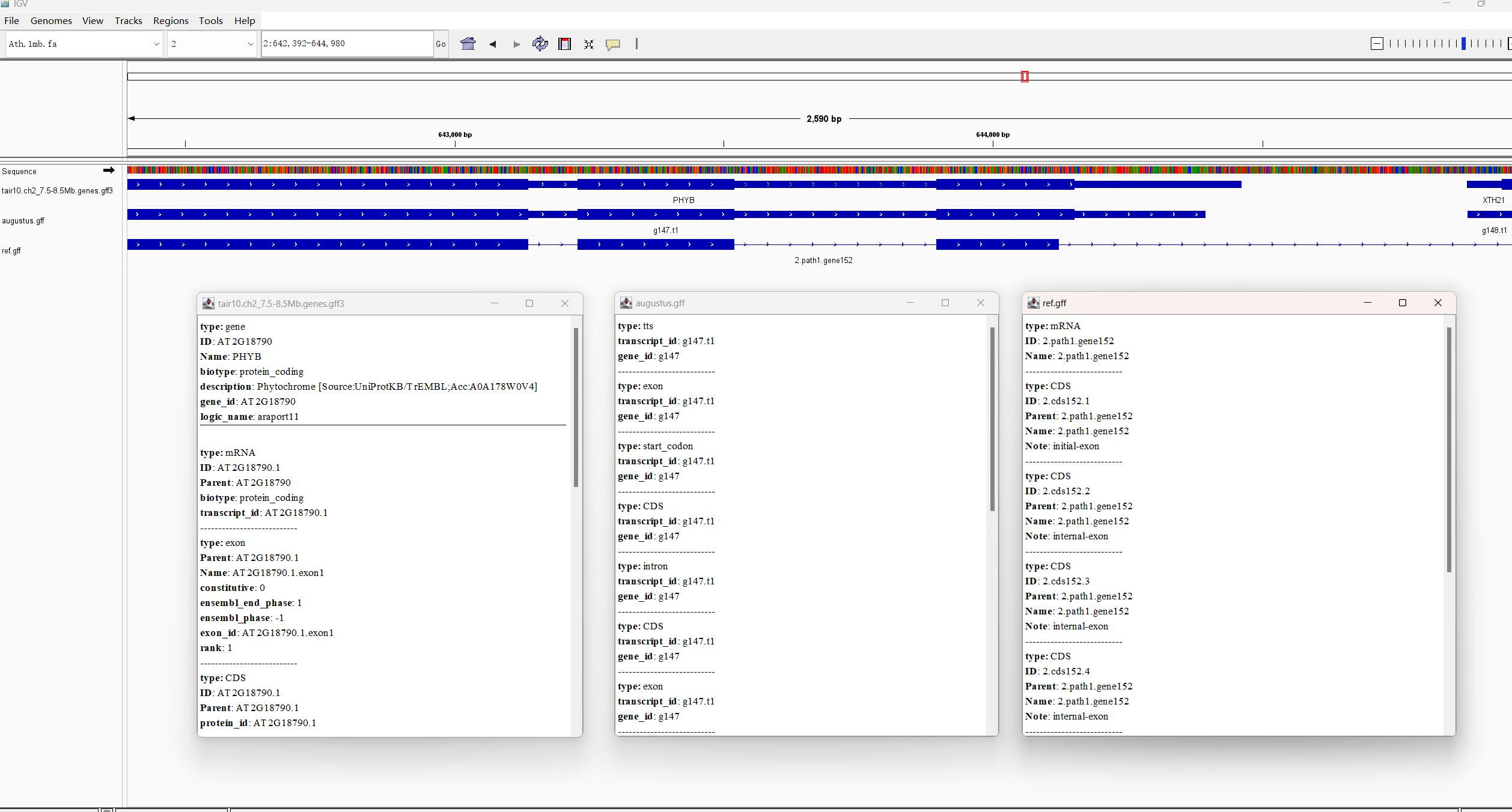
- 试列举1-2个基因在不同软件和TAIR10中的注释差异情况(结合IGV展示不同软件的注释结果)。可参考的基因:WUS、PHYTOCHROMEB
问题2.1
步骤1:使用Augustus预测
对需要使用的数据创建软连接,使用Augustus预测
1
2
3
|
cd augustus
ln -sf /home/uu02/data/ch06/Ath.1mb.fa ./
augustus --species=arabidopsis Ath.1mb.fa 1>augustus.out 2>log.txt &
|

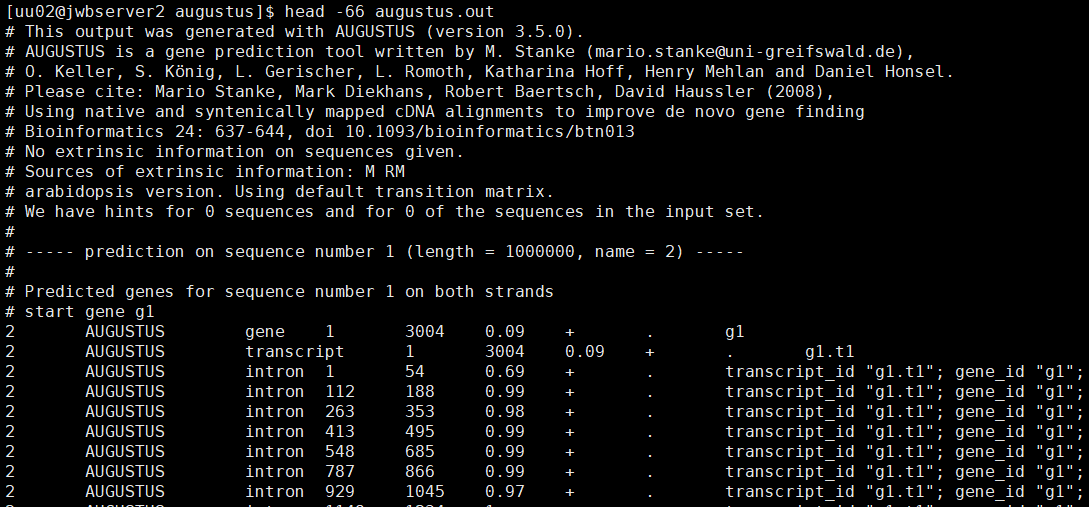
步骤2:对组装结果augustus.out统计得到基因和外显子的数量
Augusuts预测结束后得到了 augustus.out文件,这个文件本质是一个gff格式的文件,文件前半部分是一些 #开头的注释
1
2
3
4
5
6
7
8
9
|
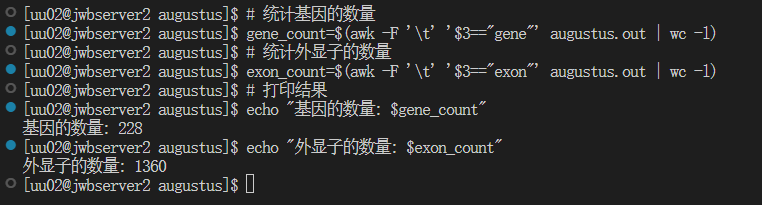
# 统计基因的数量
gene_count=$(awk -F '\t' '$3=="gene"' augustus.out | wc -l)
# 统计外显子的数量
exon_count=$(awk -F '\t' '$3=="exon"' augustus.out | wc -l)
# 打印结果
echo "基因的数量: $gene_count"
echo "外显子的数量: $exon_count"
|

**步骤3:**使用GlimmerHMM预测
对需要使用的数据创建软连接,使用GlimmerHMM预测
1
2
3
|
cd glimmerhmm
ln -sf /home/uu02/data/ch06/Ath.1mb.fa ./
glimmerhmm Ath.1mb.fa -d /home/uu02/software/07_GlimmerHMM/GlimmerHMM/trained_dir/arabidopsis -g -n 1 1>ref.gff 2>log.txt &
|

**步骤4:**对组装结果ref.gff统计得到基因和外显子的数量
组装结果也是gff文件,观察发现GlimmerHMM 的预测结果将 gene 和 exon 信息写在了 “Note” 属性中,并且 gene 信息在 “mRNA” 类型的行中
1
2
3
4
5
6
7
8
9
|
# 统计基因的数量
gene_count=$(awk -F '\t' '$3=="mRNA"' ref.gff | wc -l)
# 统计外显子的数量
exon_count=$(awk -F '\t' '$9 ~ /exon/' ref.gff | wc -l)
# 打印结果
echo "基因的数量: $gene_count"
echo "外显子的数量: $exon_count"
|

**步骤5:**选用适当的图表(如直方图)将统计结果进行展示
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
|
import matplotlib.pyplot as plt
import numpy as np
# 设置gene和extron的count
counts = {'augustus': {'gene': 228, 'extron': 1360},
'glimmerHMM': {'gene': 234, 'extron': 1245}}
# 创建堆叠的直方图
labels = list(counts.keys())
gene_counts = [counts[label]['gene'] for label in labels]
exon_counts = [counts[label]['extron'] for label in labels]
x = np.arange(len(labels)) # the label locations
width = 0.35 # the width of the bars
fig, ax = plt.subplots()
rects1 = ax.bar(x - width/2, gene_counts, width, label='gene')
rects2 = ax.bar(x + width/2, exon_counts, width, label='extron')
#加上具体的数值
for rect in rects1:
height = rect.get_height()
ax.annotate('{}'.format(height),
xy=(rect.get_x()+rect.get_width()/2, height),
xytext=(0,3), # 3 points vertical offset
textcoords="offset points",
ha='center', va='bottom')
for rect in rects2:
height = rect.get_height()
ax.annotate('{}'.format(height),
xy=(rect.get_x()+rect.get_width()/2, height),
xytext=(0,3), # 3 points vertical offset
textcoords="offset points",
ha='center', va='bottom')
ax.set_ylabel('count')
ax.set_title('gene & extron count')
ax.set_xticks(x)
ax.set_xticklabels(labels)
#图例放在表格外面
ax.legend(bbox_to_anchor=(1.05, 1), loc='upper left', borderaxespad=0.)
fig.tight_layout()
plt.show()
|

augustus文件夹下结果

部分预测结果

prodigal文件夹下结果

部分预测结果

问题2.2 不同软件预测结果的准确率和召回率
**步骤1:**使用bedtools intersectbed模块获取重叠部分符合要求的结果
1
2
3
4
5
6
7
8
9
10
11
|
#!/bin/bash
augustus='/home/uu02/02_dzx/augustus/augustus.out'
glimmerHMM='/home/uu02/02_dzx/glimmerhmm/ref.gff'
ref_data='/home/uu02/data/ch06/tair10.ch2_7.5-8.5Mb.genes.gff3'
for file in $augustus $glimmerHMM
do
dir=$(basename $(dirname $file))
intersectBed -a $ref_data -b $file -f 0.9 -r -wao > $dir.intersect
done
|
步骤2: 文件导入本地,用python统计召回率 :实际为正的样本中被预测为正样本的概率
召回率=TP/(TP+FN)
实际为正的样本数为 ref_data 参考数据中注释为gene / exon的行
实际为正 统计为真的样本,根据不同软件预测结果格式筛选
augustus: gene: data[(data[11] == 'gene') & (data[2] == 'gene')] exon: (data[11] == 'exon') & (data[2] == 'exon')]
gil mmerhmm: gene: data[(data[11] == 'mRNA') & (data[2] == 'gene')] exon: data[(data[2] == 'exon') & (data[17].str.contains('exon'))]
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
|
import os
import pandas as pd
#分别从gene、exon水平通过召回率评估不同软件预测结果
file = ['F:/生物信息软件综合实践/05_data/augustus.intersect', 'F:/生物信息软件综合实践/05_data/glimmerhmm.intersect']
ref_data = pd.read_csv('F:/生物信息软件综合实践/05_data/tair10.ch2_7.5-8.5Mb.genes.gff3', sep='\t', header=None)
ref_data_gene = ref_data[ref_data[2] == 'gene']
ref_data_exon = ref_data[ref_data[2] == 'exon']
for f in file:
data = pd.read_csv(f, sep='\t', header=None)
software = os.path.basename(f).split('.')[0]
#gene
g_p_e = len(ref_data_gene)
e_p_e = len(ref_data_exon)
if software == 'augustus':
g_tp = len(data[(data[11] == 'gene') & (data[2] == 'gene')])
g_recall = g_tp / g_p_e
e_tp = len(data[(data[11] == 'exon') & (data[2] == 'exon')])
e_recall = e_tp / e_p_e
print('augustus gene recall: %s, and turn to percentage: %s %%' % (g_recall, format(g_recall*100, '.2f')))
print('augustus exon recall: %s, and turn to percentage: %s %%' % (e_recall, format(e_recall*100, '.2f')))
#转换成百分比
elif software == 'glimmerhmm':
g_tp = len(data[(data[11] == 'mRNA') & (data[2] == 'gene')])
g_recall = g_tp / g_p_e
e_tp = len(data[(data[2] == 'exon') & (data[17].str.contains('exon'))])
e_recall = e_tp / e_p_e
print('glimmerhmm gene recall: %s, and turn to percentage: %s %%' % (g_recall, format(g_recall*100, '.2f')))
print('glimmerhmm exon recall: %s, and turn to percentage: %s %%' % (e_recall, format(e_recall*100, '.2f')))
|

| recall(召回率) |
gene |
exon |
| glimmerhmm |
12.75% |
58.35% |
| augustus |
46.61% |
68.29% |
问题2.3
列举1-2个基因在不同软件和TAIR10中的注释差异情况(结合IGV展示不同软件的注释结果)。可参考的基因:WUS、PHYTOCHROMEB


思考与讨论
1.原核生物的基因结构(组成单元、长度、GC含量等)和真核生物的基因结构有哪些差异?(结合大肠杆菌和拟南芥的注释进行分析讨论)
2.不如软件的基因预测结果,有哪些差异?为什么会有这些差异?相比于标准集,哪个软件的预测结果可能相对最好?试结合算法原理,进行讨论
1.原核生物和真核生物的基因结构差异:
- 原核生物(以大肠杆菌为例),原核生物基因没有内含子,一个基因对应一个连续的mRNA和蛋白质,大肠杆菌基因主要由连续的开放阅读框(ORF)组成,其中包含了编码蛋白质的基因。这些基因通常没有被包裹在真正的细胞核中,而是在细胞质中存在。原核生物基因整体较短,大多数大肠杆菌基因长度小于1000bp,典型的蛋白编码基因长度约为几百到几千个碱基对,原核生物基因前的启动子区域保守,存在信号序列如-10和-35元件
- 真核生物(以拟南芥为例),真核生物基因由外显子和内含子组成,外显子为编码蛋白质的区域,而内含子则是非编码序列。这些基因通常位于真正的细胞核中,即真核生物基因含有内含子,需要剪接处理长度:。真核生物基因通常较长,其中包含有复杂的调控元件和结构。蛋白编码基因的长度可能达到数千到数万个碱基对,拟南芥等真核生物基因往往很长,部分基因可达几十万bp。拟南芥等高等真核生物基因区GC含量差异较大。真核生物启动子区域结构较复杂,需多个转录因子互作才能启动转录,真核生物同属基因序列保守性差异大,难以仅依靠序列相关性预测基因
2.基因预测软件结果与实际差异的原因以及算法比较:
- 算法原理:augustus使用生成式隐马尔可夫模型,genscan和glimmerhmm使用隐马尔可夫模型
- 预测内容:augustus可以预测外显子-内含子结构,genscan和glimmerhmm仅预测外显子
因此:
- augustus利用了更丰富的外部信息,理论上预测精度应最高
- genscan和glimmerhmm仅依靠序列本身,难免会漏预测真实基因或者错误预测假基因
- 对于拟南芥这类有丰富注释的数据,以augustus训练得到的模型进行预测效果应最好
- 对于基因组注释不全的物种,三者效果相对,Glimmerhmm可能效果最好
从本次实验计算的召回率以及上述分析,总体来说,利用外部信息(如注释)训练模型进行预测的augustus,在条件允许的情况下,它的预测结果可靠性应最高。